基于人工智能技术的生物医药大数据研究在国内外均处于前沿领域,具有重要的科学价值和应用前景。国际上,美国和欧洲的顶尖研究机构在多维组学数据整合、精准医学、复杂疾病研究等方面取得了显著成就,拥有领先的技术和丰富的资源。中国在这一领域虽然起步较晚,但在国家政策支持和科研投入增加的推动下迅速发展,已在部分领域取得突破性进展。山东第一医科大学医学信息与人工智能学院孙亮教授团队开展的研究方向,主要聚焦在基于人工智能技术的多维生命组学数据整合与复杂疾病的精准分子标志物研究。

孙亮教授
孙亮教授于2013年7月在国际上首次发表了不依赖于任何已知注释信息的长链非编码RNA(lncRNA)编码潜能评估算法:CNCI(Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts)。在该研究中,孙亮教授基于大规模的多物种数据,对蛋白编码和非编码序列的组成成分,进行了深入研究,首次证明了二连体密码子的出现频率可以在跨物种的水平上用来区分蛋白编码和非编码RNA。该算法在发表的时候,虽然仅仅使用了RNA的序列特征,但是无论在实用性还是准确性等方面匀优于当时国内和国际上现有的其它方法。这一工作被Nucleic Acids Research杂志接收,申请国家专利一项,目前的引用率已经超过1600余次,在8篇CNS子刊(Nat. Med. 2018; Cell Metab. 2015,2016; Mol. Cell2022; Nat. Metab. 2021; Nat. Mach. Intell. 2019; Nat. Comun. 2021a,2021b)研究中,将其用作编码能力评分、获取候选功能实验靶点的重要手段,并且该文章也是获得2024年北京市自然科学一等奖“长链非编码基因系统发现及分子特性和功能的理论研究”,完成人(赵屹,陈润生、卜德超、孙亮、廖奇)的五篇支撑性代表作之一(图1)。


图1.北京市自然科学一等奖
为了进一步深入了解lncRNA作为一类重要的分子标志物,孙亮教授于2016年成功申报了“基于RNA绑定蛋白特异性识别序列的人类长非编码RNA分类研究”并获得国家自然科学基金青年基金的支持,目前已经结题。该项目在lncRNA一级、二级、三级序列特征分析、lncRNA与RBP结合方式的探索及非编码RNA系统性的潜在功能注释等方面取得了多个重要研究发现及成果(图2)。完成了课题目标所设定的任务。在研究过程中,孙亮教授更进一步的加深了对lncRNA序列特征的认识,特别是在以motif(k-mer)序列为代表的“标签序列”基础上对lncRNA提取了全新的分类特征,且凭借这一研究成果优化了lncRNA鉴定的分类的特征和机器学习方法,(CNIT:a fast and accurate web tool for identifying protein-coding and long non-coding transcripts based on intrinsic sequence composition),并于2019年发表在Nucleic Acids Research杂志上。该方法在小样本情况下依然可以保持良好的分类效果,针对脊椎动物群体具有更加优异的lncRNA鉴定能力,该成果加速了很多注释信息缺乏的非模式生物lncRNA的研究进展,目前总体引用达到19次,很大程度上满足了非编码RNA研究领域国内和国际上的急迫需求。
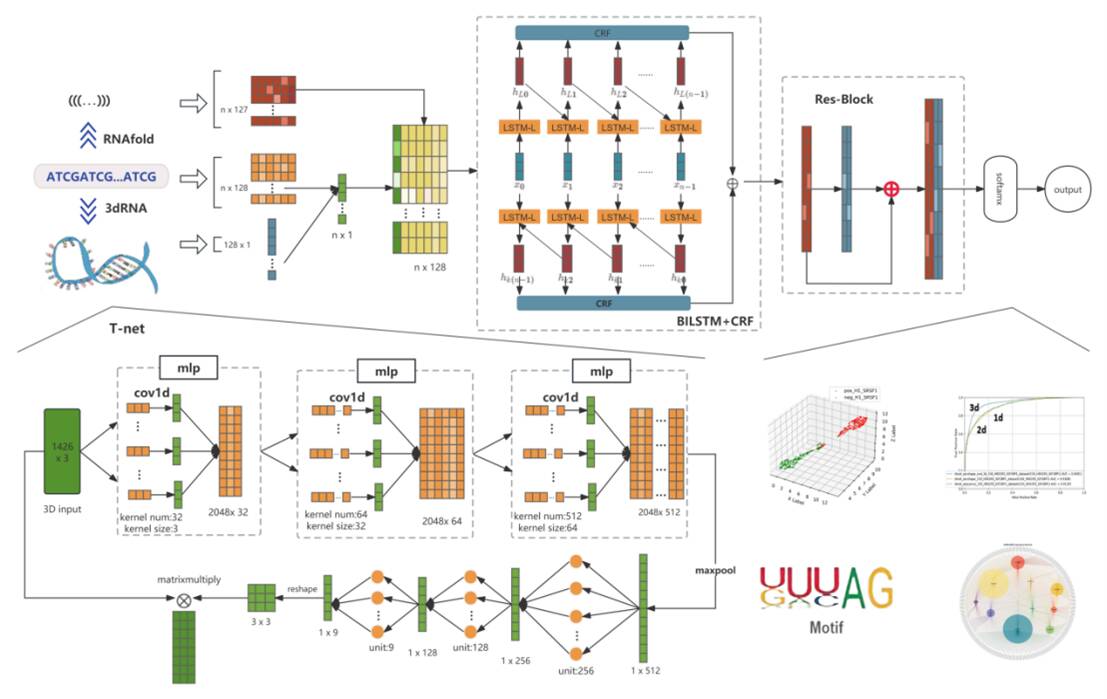
图2.非编码RNA与RNA结合蛋白(RBP)相互作用预测算法(NeuroPRIS)流程
在lncRNA序列特征和人工智能鉴定算法研究取得显著科研成果的基础之上(CNCI,CNIT),进一步整合了目前国际上最权威的公开数据资源,并运用大量统计分析方法发现了这些人类可编码lncRNA及其编码序列的独有特征,并以此为依据构建了一个全新的序列评估矩阵和打分体系,该方法可以将lncRNA中的编码序列、mRNA的ORF序列以及真正的非编码序列进行很好的区分。本项研究于2020年,获得国家自然科学基金面上项目《长链非编码RNA中的短肽序列鉴定与特征研究》的资助,在该项目支持周期内,研究团队基于动态规划与深度学习算法,开发了多个可翻译lncRNA及环状RNA的预测算法及数据库平台,目前有两篇高水平研究成果均于2024年发表在Nucleic Acids Research杂志上CICADA: (a circRNA effort toward the ghost proteome)、LncPepAtlas: ( a comprehensive resource for exploring the translational landscape of long non-coding RNAs),并成功申请国家发明专利两项(图3、图4)。且上述算法大规模鉴定得到的一系列非编码分子标志物目前已经全面进入功能机制验证阶段。
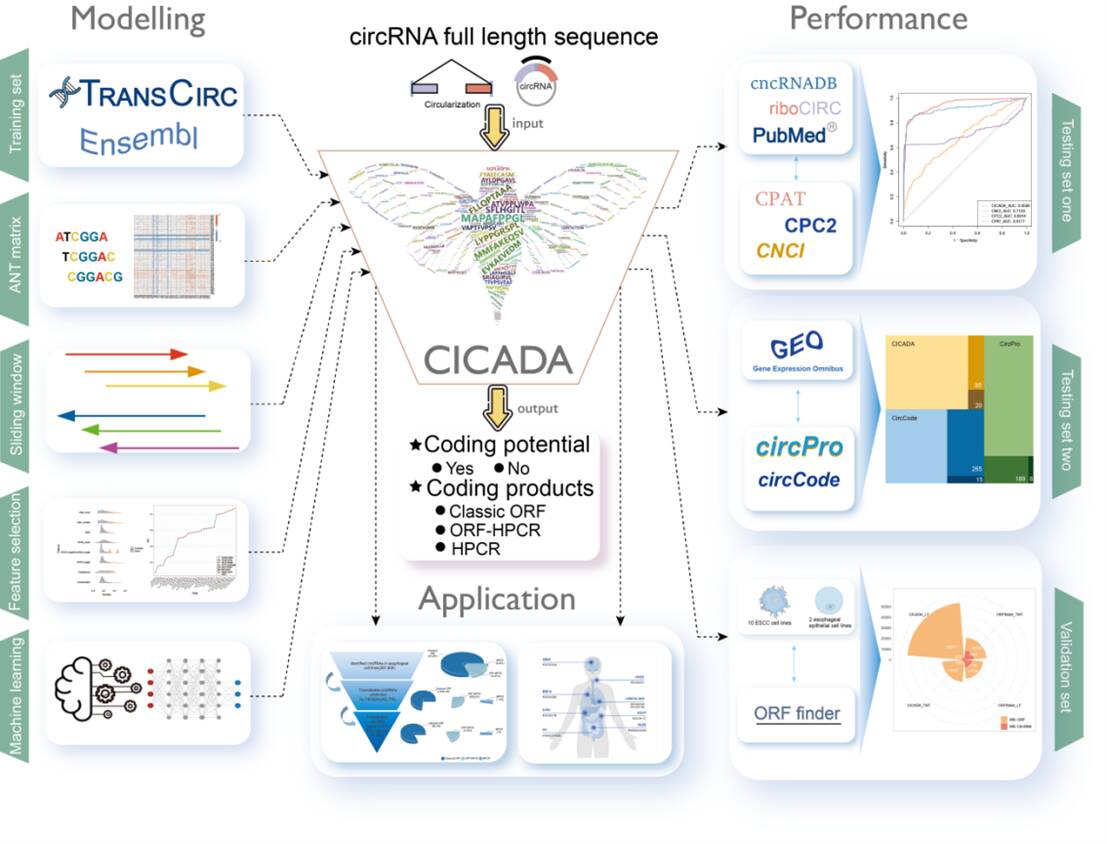
图3. 人类可翻译circRNA预测算法全流程
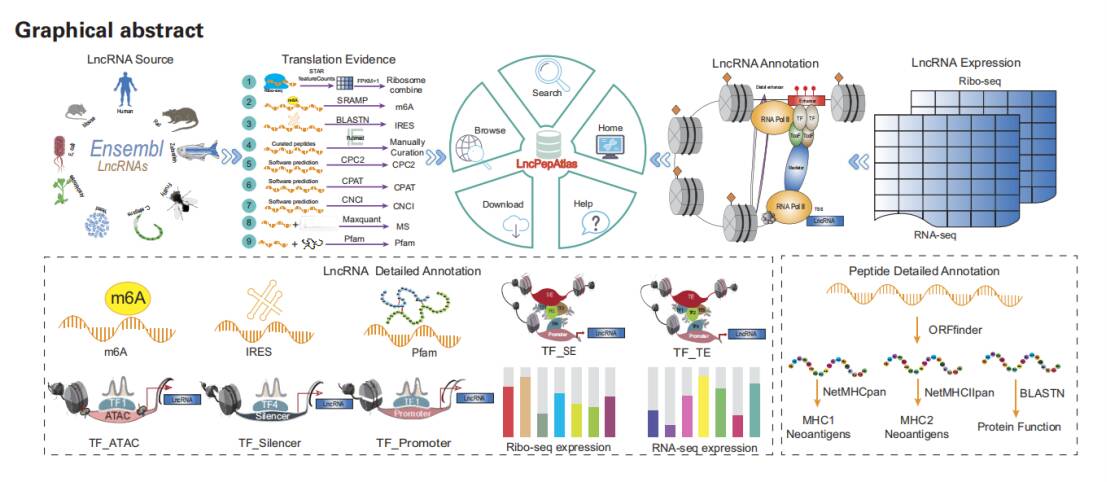
图4. 人类可翻译非编码RNA综合注释平台
近两年,孙亮教授研究团队使用大语言模型和生成式人工智能的技术开发了一个基于人工智能技术的药物测算大平台,从多组学数据中系统挖掘现有药物与疾病的潜在关联,旨在为药物靶点识别及新的适应症发现提供科学依据,并且进一步结合上述我们在分子标志物中的研究基础,对有明确靶点但是缺乏治疗药物的疾病,采用生成式的人工智能技术进行药物从头设计(图5)。
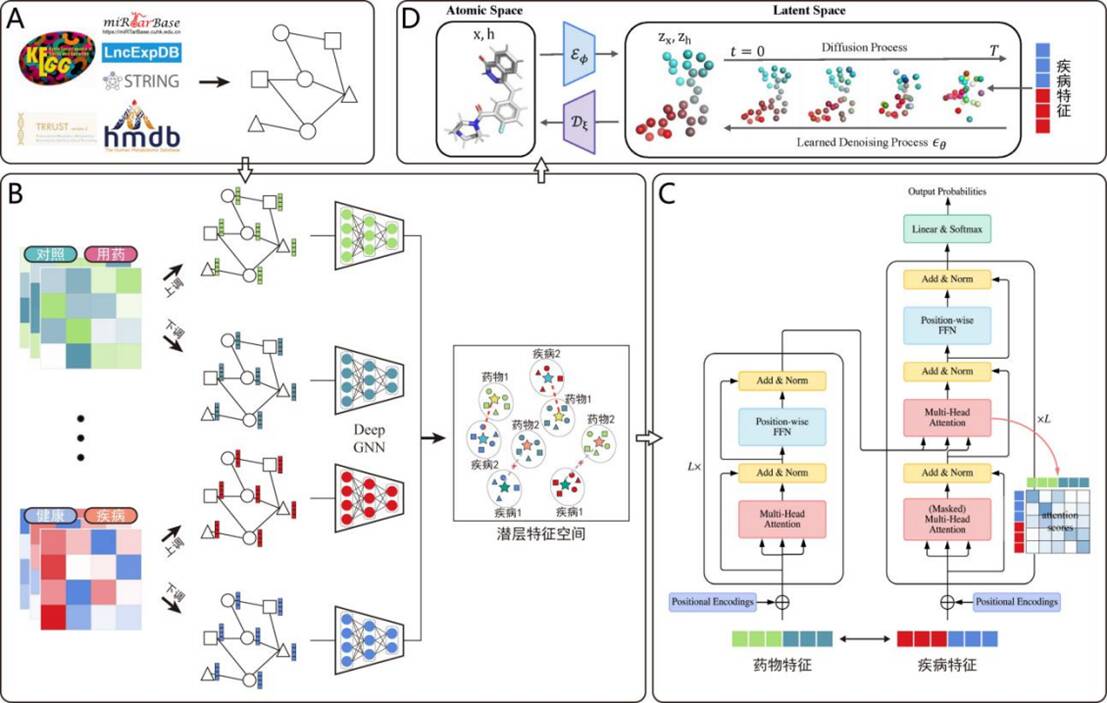
图5. AIDdrug的模型框架:药物重定位、药物靶点识别以及从分子结构层面重新设计疾病的潜在治疗药物
该项研究目前已经和某企业签订了总价500万元的技术开发合同,分为以下三个主体研究内容:《基于人工智能技术的药物测算平台》、《基于人工智能技术的类器官培养体系》、《基于类器官及人工智能技术的创新药物开发》。



