山东第一医科大学医学信息与人工智能学院孙亮教授团队在长链非编码RNA翻译小蛋白方面取得研究进展,相关成果以“LncPepAtlas:探索长链非编码RNA翻译景观的综合资源(LncPepAtlas: a comprehensive resource for exploring the translational landscape of long non-coding RNAs)”为题,于2024年10月22日在线发表于《Nucleic Acids Research》(IF=16.6)杂志,论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkae905/7831081。
长非编码RNA(lncRNAs)曾被传统上视为基因表达的非编码元件,但随着分子生物学研究的深入,科学界逐渐揭示了它们在多种生物学过程中的关键作用。lncRNAs不仅参与基因表达的调控,而且有些lncRNAs能够翻译成微肽,在细胞分化、信号传导、代谢调节等多种细胞过程中发挥功能。随着生物技术和计算算法的进步,研究者们引入了一系列新方法来研究lncRNAs的翻译机制。然而目前的lncRNAs翻译的证据比较单一,因此,迫切需要提供对lncRNAs上游调控的全面注释以及整合跨物种lncRNAs编码微肽翻译证据的多种来源的资源。
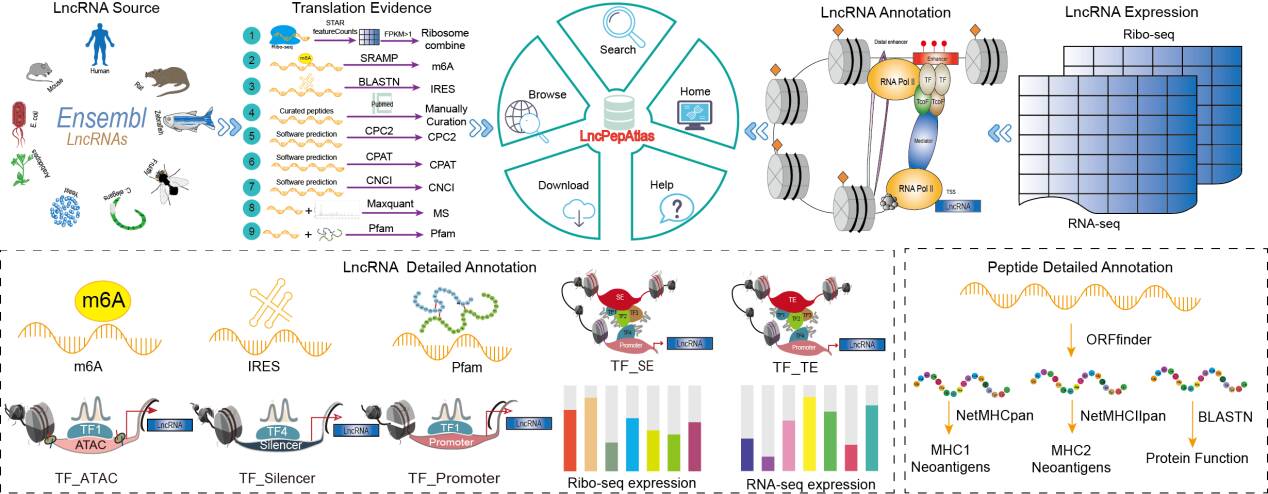
LncPepAtlas数据库整合了来自九个不同来源的lncRNAs翻译的强有力证据,其中包括收集并分析包含来自9个物种的2631个公开可用Ribo-seq样本的数据集。涵盖人、小鼠、大鼠、拟南芥、秀丽隐杆线虫、黑腹果蝇、斑马鱼、大肠杆菌和酿酒酵母。并采用了九种方法来评估Ensembl数据库中lncRNAs衍生微肽的翻译证据,包括MC、Riboseq、IRES、m6A、MS、Pfam、CPC2、CPAT和CNCI。除了探索翻译证据外,LncPepAtlas还为lncRNAs提供了广泛的注释,旨在识别上游调控注释,如转录因子(TF)、超级增强子(SE)和增强子(TE),以及各种癌症类型、组织或细胞系的lncRNAs表达注释。此外,LncPepAtlas还分析了lncRNAs编码的微肽,包括它们与实验验证的蛋白质序列的比对,并评估了它们作为与I类和II类主要组织相容性复合体(MHC)分子相互作用的抗原肽的结合亲和力。LncPepAtlas提供了一个直观的界面,便于搜索、浏览和下载可翻译lncRNAs及其编码微肽的全面细节。
LncRNAs翻译小蛋白资源构建